Nomogram文献分析:提取数据
前言
今天教大家如何分析Nomogram类型的文章,并提取相应的MIMIC-III数据。
要分析的文章:https://pubmed.ncbi.nlm.nih.gov/36504658/ 。这是一篇典型的mimic-iii数据分析的套路,构建一个Nomogram预测某种结局发生的影响因素。

这个研究旨在调查全身免疫炎症(SII)与颅内出血(ICH)患者在重症监护病房(ICU)的住院死亡风险之间的关联,并进一步开发与SII相关的预测模型,以预测ICH患者的住院死亡风险。
在训练组中,232名ICH患者死亡,708名存活。LASSO回归显示一些预测因子,包括白细胞计数、血糖、血尿素氮、SII、格拉斯哥昏迷评分、年龄、心率、平均动脉压、红细胞、碳酸氢盐、红细胞分布宽度、肝硬化、呼吸衰竭、肾功能衰竭、恶性肿瘤、血管加压药和机械通气。建立了整合这些预测因子的预测模型。在训练组中,量表图下面积(AUC)为0.810,在测试组中为0.822,表明这个量表图可能有良好的性能。
结论:全身免疫炎症与ICU中ICH患者的住院死亡风险增加有关。已开发并验证了一种针对ICU中ICH患者的住院死亡风险的量表图。
第一步:文章思路分析
1. 病人对象 Subject
要提取数据,你首先必须知道自己的研究对象是什么。这篇文章里,研究对象是颅内出血的病人,那么哪些是这些病人呢?需要通过ICD-9、ICD-10进行判断了。文章里面的ICD用的是:

2. 入组标准
所以入组的研究对象是ICD-9 code是:431, 432, 9487, 77210-77214。首先就要把这些病人挑出来。
3. 排除标准
(1) aged <18 years;
(2) were hospitalized in the ICU for <24 h;
(3) had missing data of SII; and
(4) had missing data of other covariates.
4. 终点指标
你需要知道你要分析的结局指标是啥,这是文章的落脚点。常说的Y。这篇文章里面就是指的住院死亡 in-hospital death。
5. 独立因素
就是病人相关的指标了,一般包括基础资料,血氧血气,生化,vital sign,并发症,ICU 评分等等了。一般Table 1都会列出的了,这篇文章大致是:

6. 分析过程
这个是一般的数据处理过程,大致为:LASSO (单因素分析)--》 多因素逻辑回归 --》Nomogram 构建 --》Nomogram的评价,如ROC/AUC,c-index,IDI, NRI, DCA等等。比如另一篇文章的比较全面的流程图:
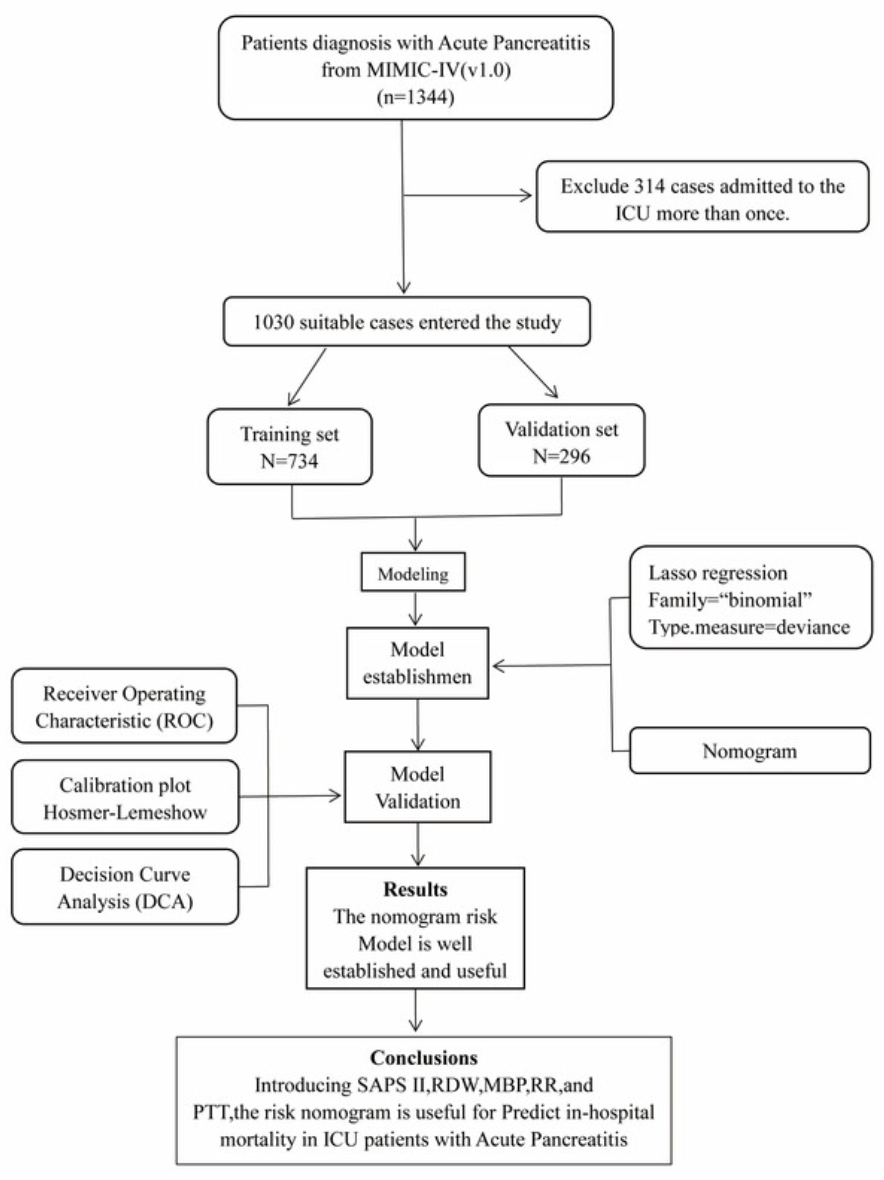
好了,下面我们开始进行提取数据,并尝试重新这篇简单文章的Nomogram的构建。
第二步:提取数据
其实系统已经设计得很简单,零代码,只需要点点勾勾就能导出你要的数据。一般来讲,大家提取衍生库的内容都够用了,原生库的原始数据一般很粗糙,没有归类总结。比如MIMIC-III数据库的chartevents,患者的住院期间的图表信息,数据量庞大,也是MIMIC-数据库臃肿的原因。labevent也是个大表,但里面其实我们需要的就是些血氧血气生化指标,大部分都在衍生库里面。除非你有些特殊要求,不建议提这个数据。
1. 输入ICD code,选择病人队列
如前面提到的,进入MIMIC-III数据库,点击“ICD搜索”,选择‘Disease’,选择‘全部诊断’ (就是不区分是否第一诊断),搜索框里面输入 431, 432, 9487, 77210, 77211, 77212, 77213, 77214 (多个icd code逗号区分),当然也可以输入关键词,比如 “intracerebral hemorrhage”。所有包含这些字符的ICD code都会显示,找到你想要的,进行勾选,注意 可跨页勾选!
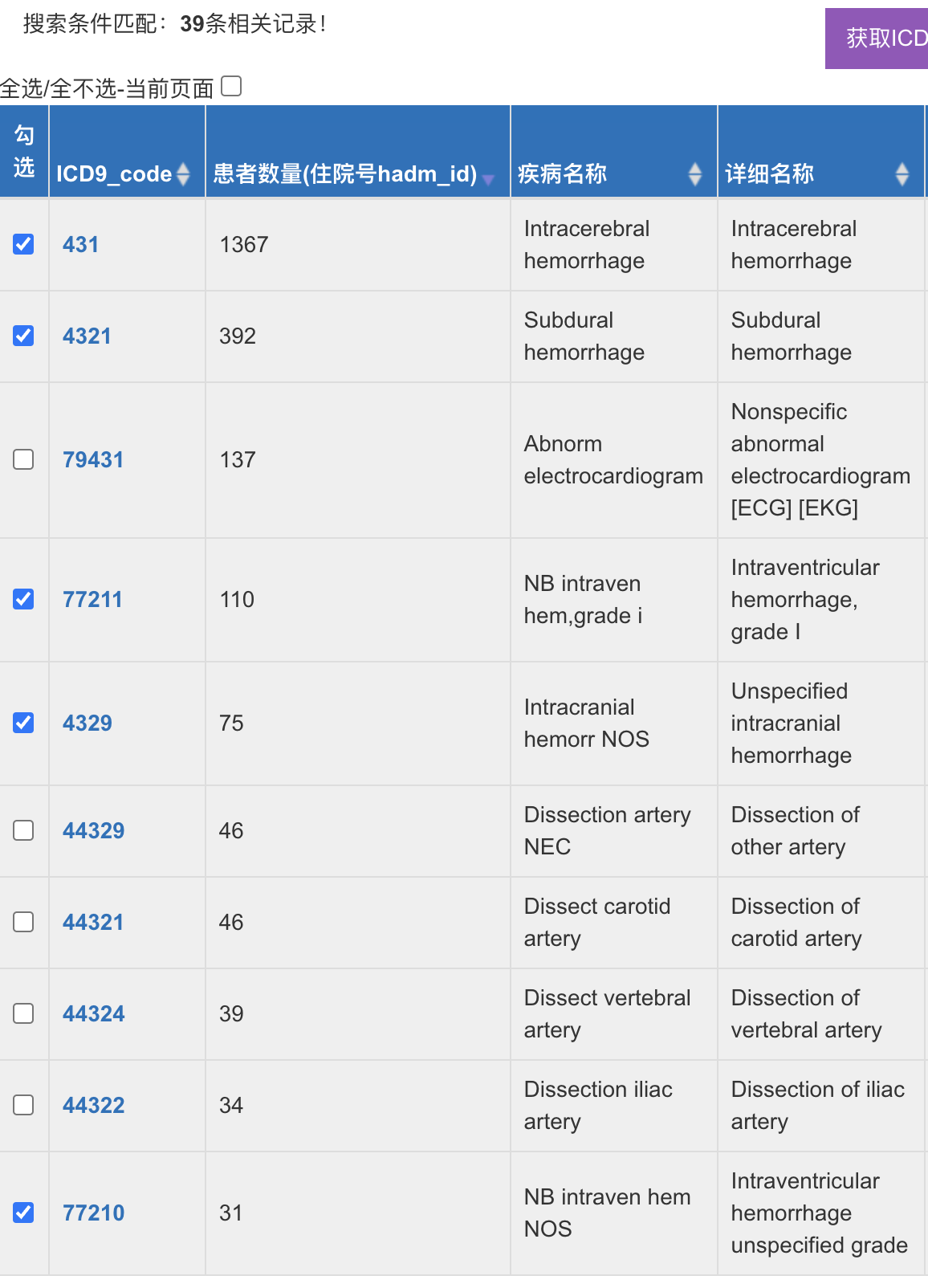

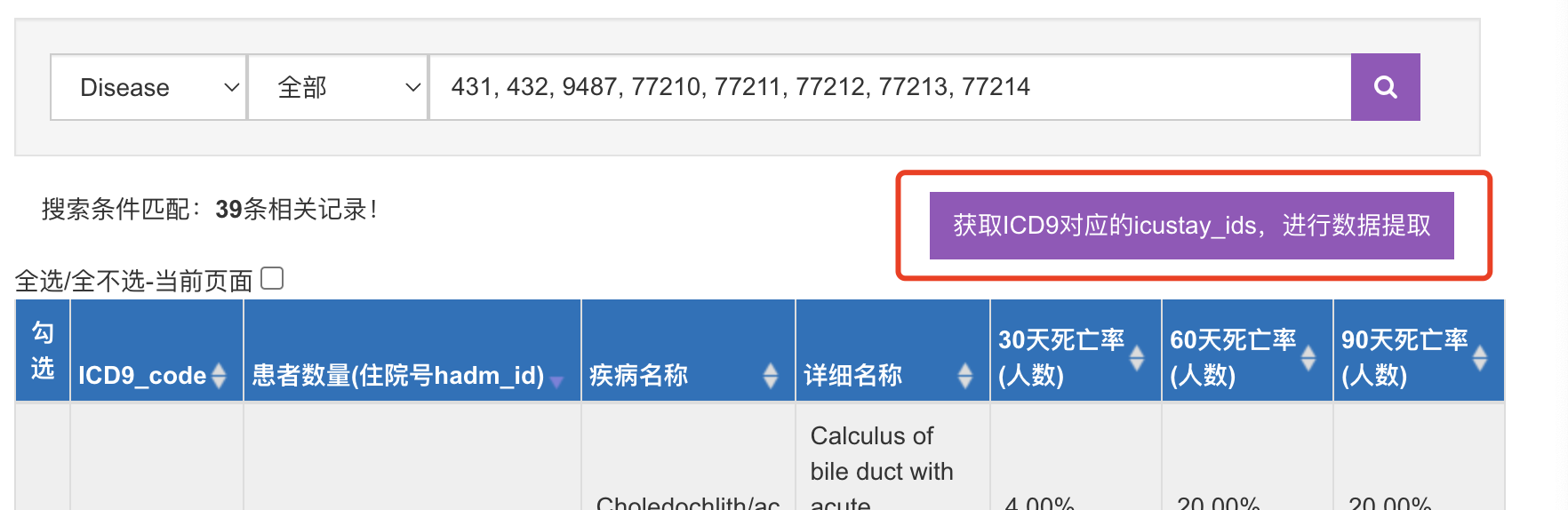
虽然系统会直接跳到数据提取页面,但是在“输入icustay_ids”这里可以看到你勾选的所有病人的icustay_id:
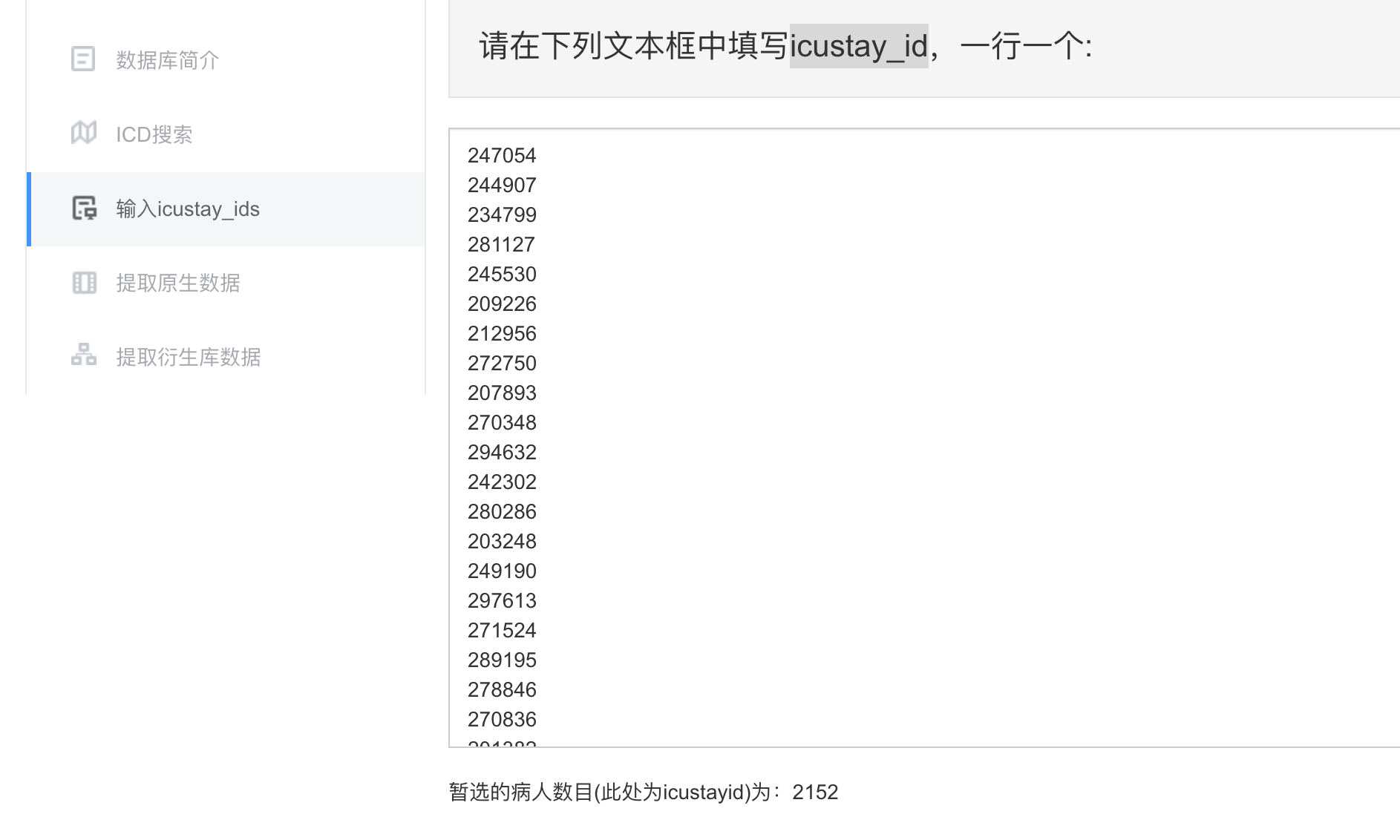
做这个输入,是为了给你提供方便,你如果知道自己icustay_id,直接清空之后粘贴,然后点击 “提交icustayid,开始提取数据 ”就可以提取你的病人队列了。文章是1925个,这里有2152个,可能和我选择的ICD code有关,比如我这里勾选了蛛网膜下出血。当有多个ICD code的时候,一定要设置准确!
2. 导出数据
这个很简单了。一个个指标勾选,导出数据即可。其实我们的数据主要在“衍生库”里面:
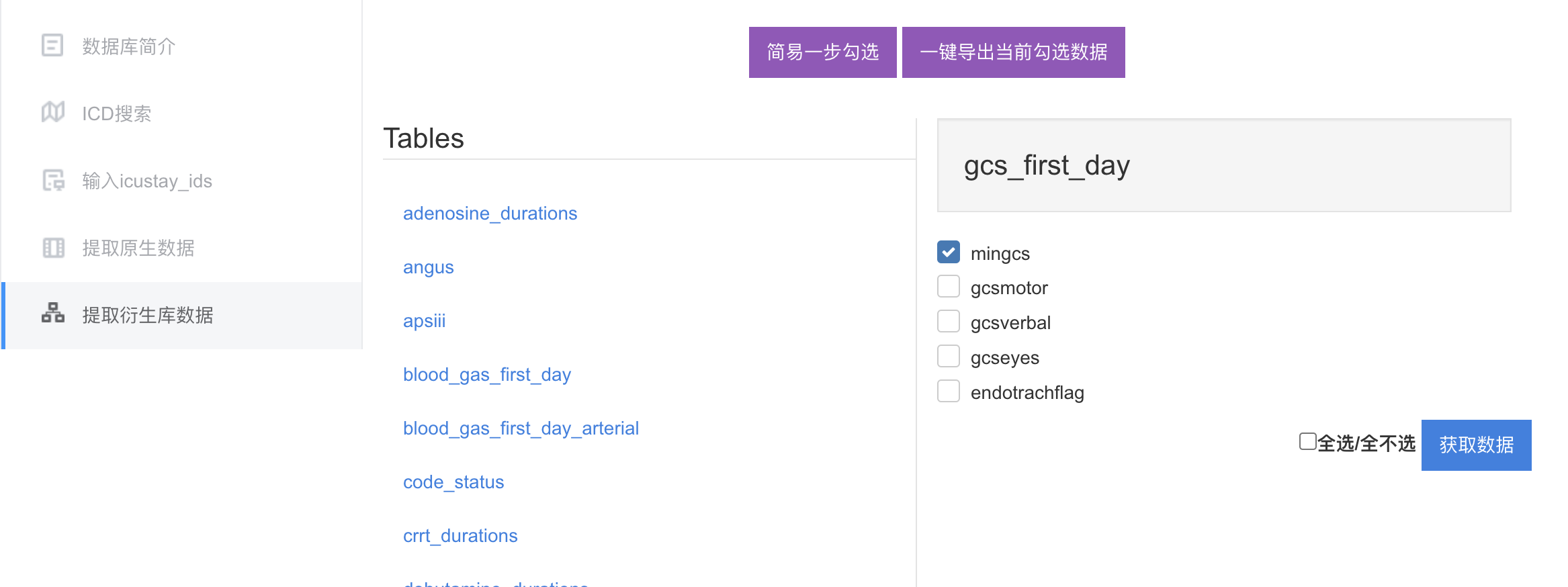
如果你不知道这些指标叫什么,建议还是多补充背景知识。
如果偷懒些,我帮你预置了一些指标,一步勾选,一键导出即可。
原生数据库,我们只需要病人资料 patients 表,和adminssion表的内容即可。用于后面判断是否住院死亡。
下面是两分钟就提好的这个文章的数据了:
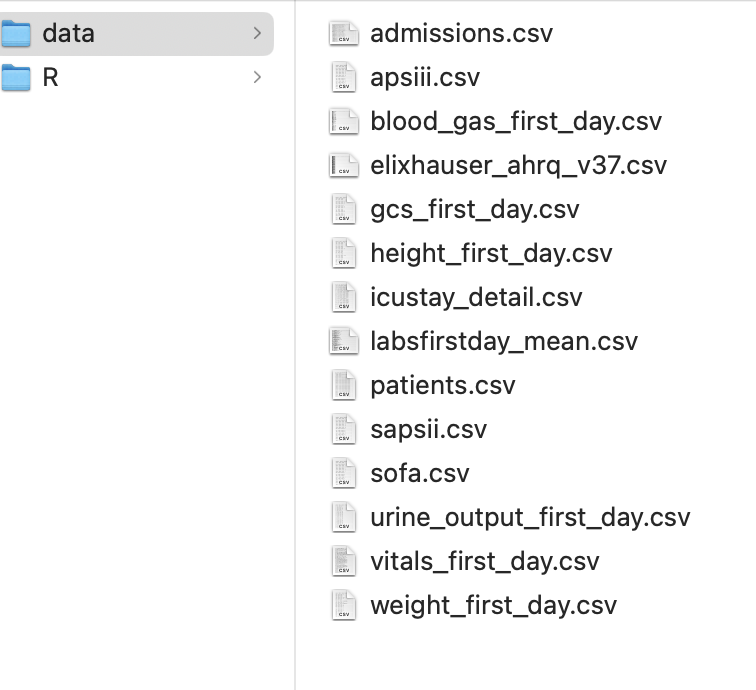
每个csv都是 tab分格的数据,如果用Excel打开会显示挤到一起,需要手动导入分割才能看到。一般我们都是在R里面进行后续处理的了。csv文件,直接当txt打开,长这样:
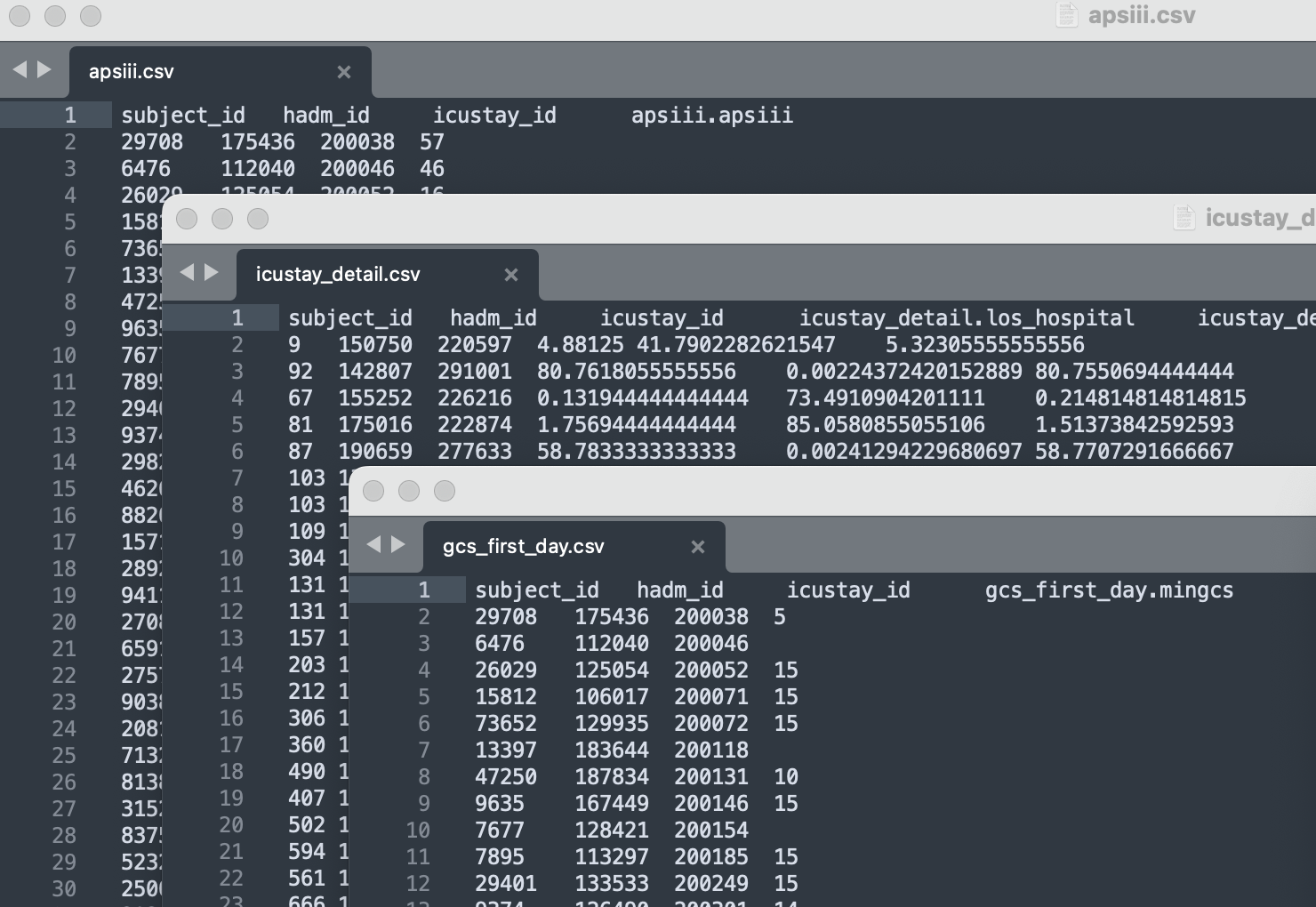
GCS这个指标对出血病人还是挺重要的。
这样,数据导出就完成了,总共花费了10分钟不到。
下面开始数据合并和处理,建模分析。
第三步:数据预处理
大致分为几个步骤:1)数据合并;2)病人队列排除;3)指标缺失值处理;4)变量初始化。
1. 数据合并
为什么不推荐对所有表进行简单的连接合并?因为你会看到这些表不是所有字段唯一的。
数据导入R里面,先看看数据行数在每个表的情况:
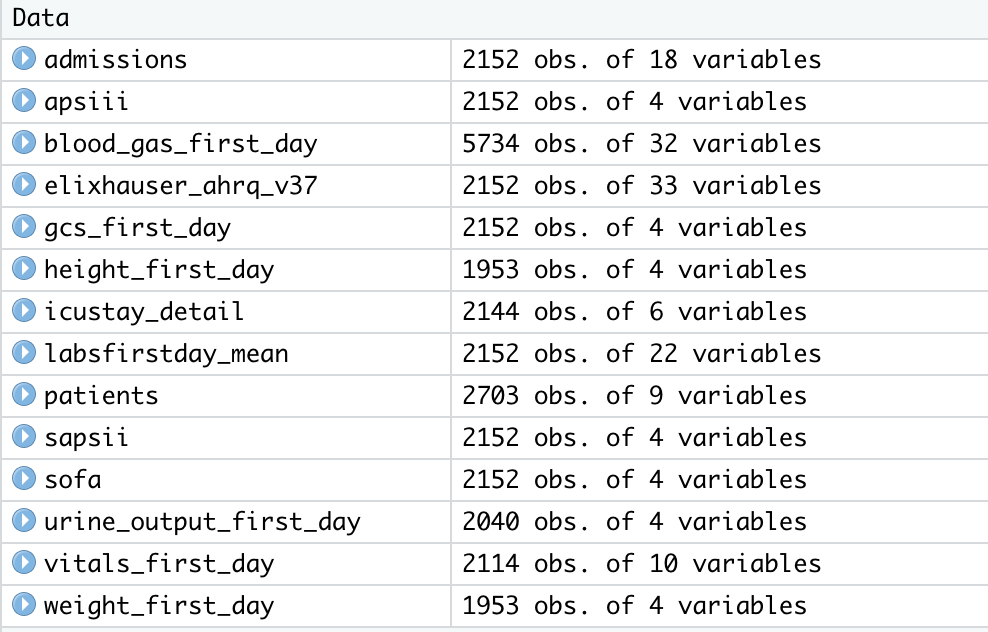
大部分是2152行数据,和前面的数据icustay_id 数量相符。但血气值有5k多行,patients有2703行,icu_stay只有2144行。而有些数据有缺失,比如vital sign只有2114行。以哪一个表为起始表进行合并要根据实际情况就行。
我这里以admission为起始进行合并,结果如下:
> dim(merge.data) [1] 2152 94 # 合并之后又2152个队列,有94个指标
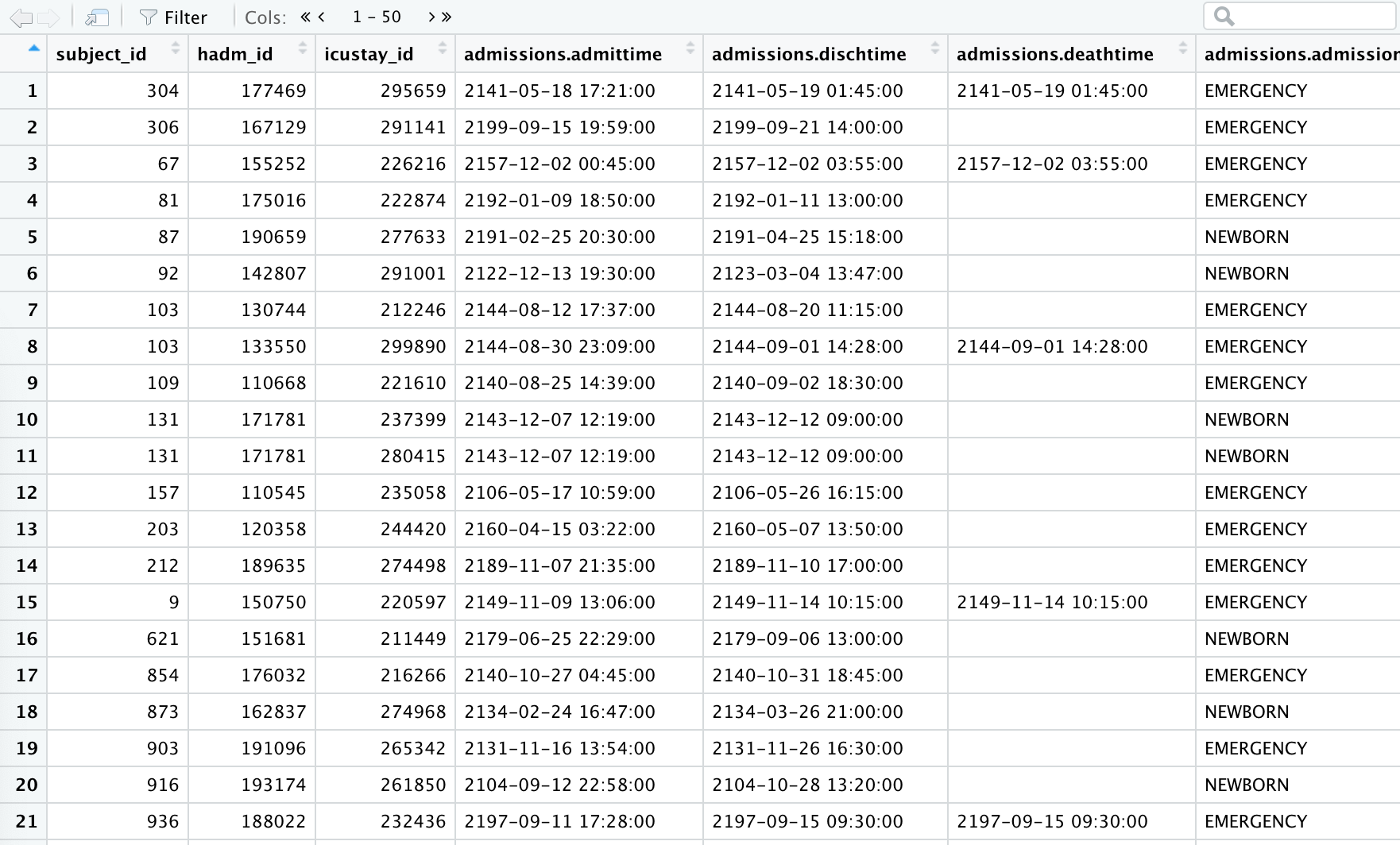
其中住院死亡是结局:admissions.hospital_expire_flag 表里面直接给出了。
2. 排除病人
标准:1) age < 18; 2) icu 时长超过24 小时
### exclude age less than 18 raw.ready <- raw.ready[raw.ready$icustay_detail.admission_age > 18,] ### exclude patients less than 24 hours raw.ready <- raw.ready[raw.ready$icustay_detail.los_icu>=1,]
3. 缺失值处理
看看缺失情况:
缺失大于20%的指标需要删除:
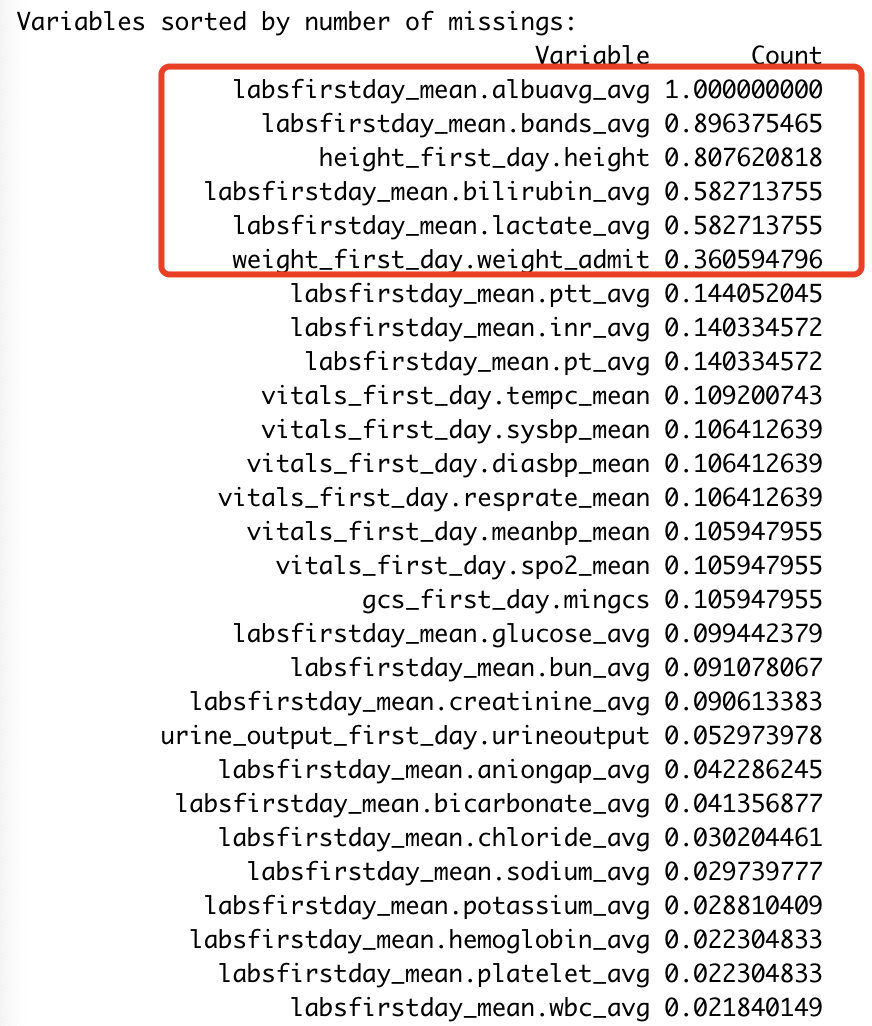
然后还需要对缺失值进行插值补全。这样数据基本准备好了。可以开始建模进行后续分析了。至此不再赘述。
第四步:欣赏下我们用这个系统做出来的图
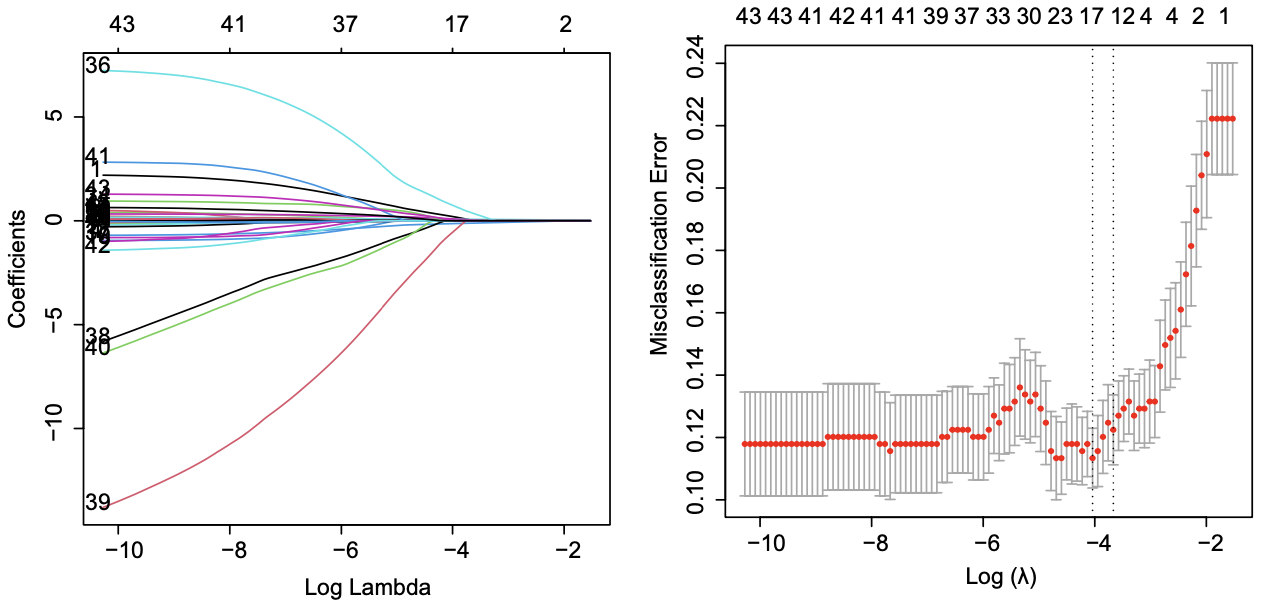
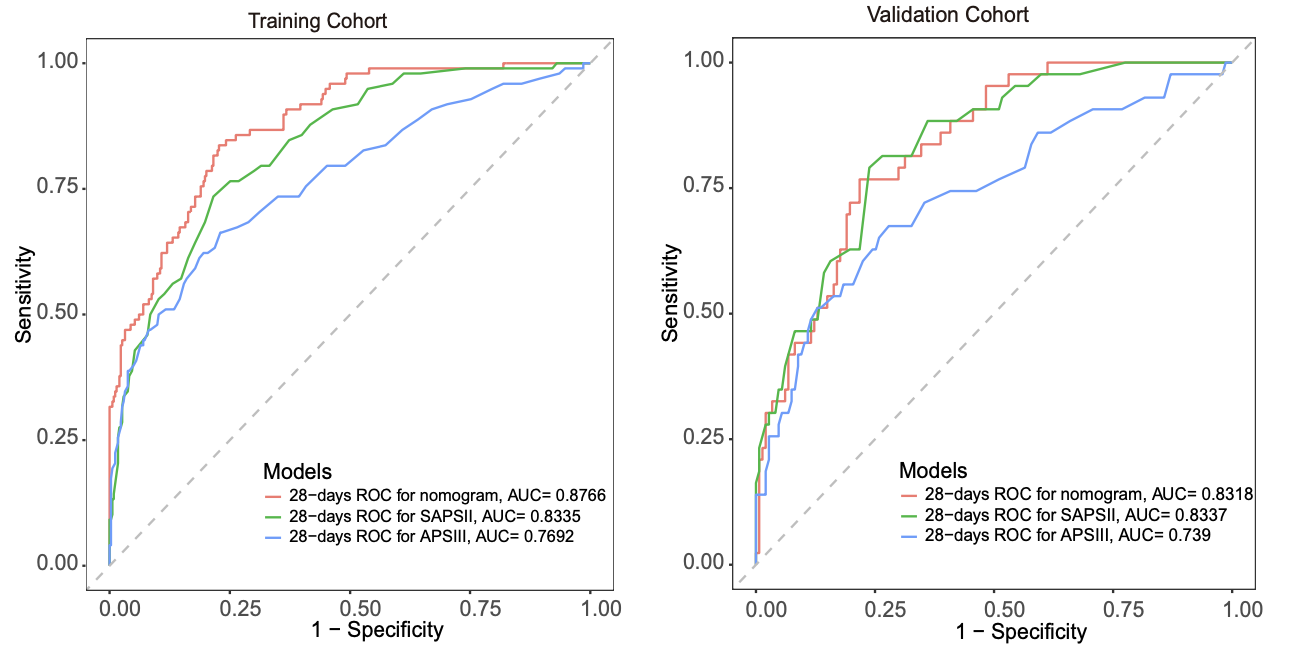
最后
另外一些功能也还在陆续开发中,敬请期待。